VMC
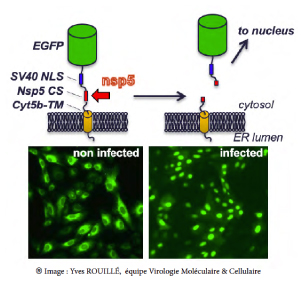
Structure d'une sonde fluorescente utilisée pour révéler l'infection par le virus SARS-CoV-2. La sonde fluorescente est constituée d'une protéine fluorescente verte (EGFP) à laquelle sont fusionnés un site de localisation nucléaire (SV40 NLS), un site de clivage protéolytique spécifique de la protéase nsp5 du SARS-CoV-2 (Nsp5 CS) et le domaine transmembranaire de la protéine cytochrome 5b (cyt5b-TM). Lorsqu'elle est exprimée dans une cellule non infectée, cette sonde est localisée dans le cytoplasme par son domaine transmembranaire. Lorsque la cellule est infectée, la protéase virale clive la sonde, ce qui libère la GFP de son ancrage membranaire et elle est alors dirigée vers le noyau de la cellule. Ainsi, les cellules non-infectées présentent une fluorescence cytoplasmique, alors que dans les cellules infectées, la fluorescence est nucléaire.
Łoczechin A, Séron K, Barras A, Giovanelli E, Belouzard S, Chen YT, Metzler-Nolte N, Boukherroub R, Dubuisson J, Szunerits S. Functional Carbon quantum dot as medical countermeasures to human coronavirus (HCoV). ACS Applied Materials & Interfaces, 2019, 11, 42964-42974
Cantrelle FX, Boll E, Brier L, Moschidi D, Belouzard S, Landry V, Leroux F, Dewitte F, Landrieu I, Dubuisson J, Deprez B, Charton J, Hanoulle X. NMR spectroscopy of the main protease of SARS-CoV-2 and fragment-based screening identify three protein hotspots and an antiviral fragment. Angew Chem Int Ed Engl, 2021, 60, 25428-25435
Meunier T, Desmarets L, Bordage S, Bamba M, Hervouet K, Rouillé Y, François N, Decossas M, Tra Bi FH, Lambert O, Dubuisson J, Belouzard B, Sahpaz S, Séron K. A photoactivable natural product with broad antiviral activity against enveloped viruses including highly pathogenic coronaviruses. Antimicrob Agents Chemother, 2022, 66, e01581-21
Desmarets L, Callens N, Hoffmann E, Danneels A, Lavie M, Couturier C, Dubuisson J, Belouzard S, Rouillé Y. A Reporter Cell Line for the Automated Quantification of SARS-CoV-2 Infection in Living Cells. Front Microbiol, 2022, 13, 1031204
Brie L, Hassan H, Hanoulle X, Landry V, Moschid D, Desmarets L, Rouillé Y, Dumont J, Herledan A, Warenghem S, Piveteau C, Carré P, Ikherbane S, Cantrelle FX, Dupré E, Dubuisson J, Belouzard S, Leroux F, Deprez B, Charton J. Novel dithiocarbamates selectively inhibit 3CL protease of SARS-CoV-2 and other coronaviruses. Eur J Med Chem, 2023, 250, 115186
Equipe fondatrice
Equipe Virologie Moléculaire & Cellulaire
Dans le cadre du projet ViroCrib, l’équipe VMC offre la possibilité de cribler des chimiothèques par une approche à haut contenu (HCS) sur des cellules infectées par le SARS-CoV-2 en niveau de confinement 3 grâce à son accès à la plateforme ARIADNE-criblage de l’UAR Plateformes Lilloises en Biologie et Santé (UMS-2014) localisée sur le campus de l’Institut Pasteur de Lille (UMS-PLBS). Cette plateforme est constituée de trois plateaux. Le plateau HTS permet de cribler des librairies chimiques à haut débit. Les plateaux HCS-L2 et HCS-L3 permettent de réaliser à haut débit et à haut contenu des criblages par microscopie automatisée et quantitative. . De façon plus large, l’équipe VMC apporte son expertise dans le domaine de l’étude des coronavirus. Elle contribue à l’identification et la caractérisation d’antiviraux et elle met au point différents outils complémentaires au criblage et à la caractérisation de l’activité antivirale de composés identifiés. Elle a notamment mis au point un outil de criblage basé sur la translocation nucléaire d’une protéine fluorescente suite à l’infection par le SARS-CoV-2, permettant d’effectuer rapidement des criblages à haut débit. Cette approche est transposable à d’autres modèles viraux.